 Científicos estadounidenses del Departamento de Energía, del Joint Genome Institute (JGI) y de la Universidad de Carolina del Norte han desarrollado un catálogo de genomas bacterianos para ayudar a otros investigadores a identificar y caracterizar genes candidatos que ayuden a las bacterias a prosperar en ambientes vegetales, especialmente involucrados en la colonización de raíces bacterianas.
Científicos estadounidenses del Departamento de Energía, del Joint Genome Institute (JGI) y de la Universidad de Carolina del Norte han desarrollado un catálogo de genomas bacterianos para ayudar a otros investigadores a identificar y caracterizar genes candidatos que ayuden a las bacterias a prosperar en ambientes vegetales, especialmente involucrados en la colonización de raíces bacterianas.
“Si queremos diseñar el microbioma correcto para apoyar el crecimiento de las plantas, necesitamos entender la función real del microbioma y no solo los genes marcadores de secuencia”, explica el coautor del estudio Asaf Levy, investigador científico GJI. “Aquí usamos un esfuerzo genómico y computacional masivo para abordar la pregunta fundamental e importante: ¿Cómo interactúa el microbioma de la planta con la propia planta?”
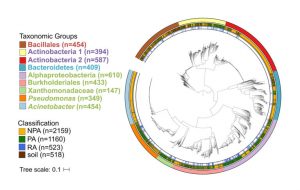
Científicos de varias instituciones han aislado previamente nuevas bacterias del entorno de la raíz de Brassicaceae (191), álamos (135) y maíz (51). Los genomas de estos 377 aislamientos bacterianos, además de 107 células bacterianas individuales adicionales de las raíces de Arabidopsis, se secuenciaron, ensamblaron y anotaron en el JGI.
Después, los investigadores de combinaron los nuevos genomas con los genomas disponibles públicamente que representan los principales tipos de bacterias vinculadas a las plantas, así como las de los ambientes no vegetales. Los datos combinados llevaron a la formación de la base de datos con 3.887 genomas, en donde 1.160 eran de plantas.
[FUENTE: JGI]













